library(sf)Linking to GEOS 3.11.0, GDAL 3.5.3, PROJ 9.1.0; sf_use_s2() is TRUEIn questa lezione vedremo:
come interpolare su una griglia regolare di punti un fenomeno continuo nello spazio e osservato in un numero limitato di osservazioni
come creare un documento di tipo RMarkdown
I pacchetti utilizzati sono sf (già utilizzato in Chapter 8 per la gestione di oggetti spaziali e la loro rappresentazione)
library(sf)Linking to GEOS 3.11.0, GDAL 3.5.3, PROJ 9.1.0; sf_use_s2() is TRUEe gstat. Quest’ultimo è da installare, secondo quanto descritto in Section 4.2, verrà poi utilizzato per la procedura di interpolazione spaziale
library(gstat).RDataPer questa parte utilizzeremo il file ariasf.RData disponibile nella cartella Dati della pagina Moodle del corso. Un file con estensione .RData è un file di dati creato dentro R e poi esportato (è un formato proprietario di R). Ora per importare i dati contenuti in ariasf.RData usiamo la funzione load (l’unico input da dare è la posizione del file all’interno del proprio computer; eventualmente utilizzare la funzione file.choose() descritta in Section 8.1):
load("./data/ariasf.RData")La precedente funzione carica nell’ambiente di lavoro un oggetto di nome ariasf che risulta essere un oggetto spaziale (ovvero un data frame caratterizzato da una geometria, si veda anche Chapter 8):
library(tidyverse)── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.4
✔ forcats 1.0.0 ✔ stringr 1.5.0
✔ ggplot2 3.5.1 ✔ tibble 3.2.1
✔ lubridate 1.9.2 ✔ tidyr 1.3.0
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorsclass(ariasf)[1] "sf" "tbl_df" "tbl" "data.frame"Le variabili contenute in ariasf sono le seguenti
colnames(ariasf)[1] "IDStations" "mediapm" "geometry" ovvero l’identificativo di ogni stazione, media del PM10 per l’anno considerato insieme alle informazioni sulle coordinate (geometry) delle 106 stazioni di monitoraggio.
Rappresentiamo graficamente, tramite un istogramma, la distribuzione della variabile mediapm:
ariasf %>%
ggplot() +
geom_histogram(aes(mediapm),
col="white")`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.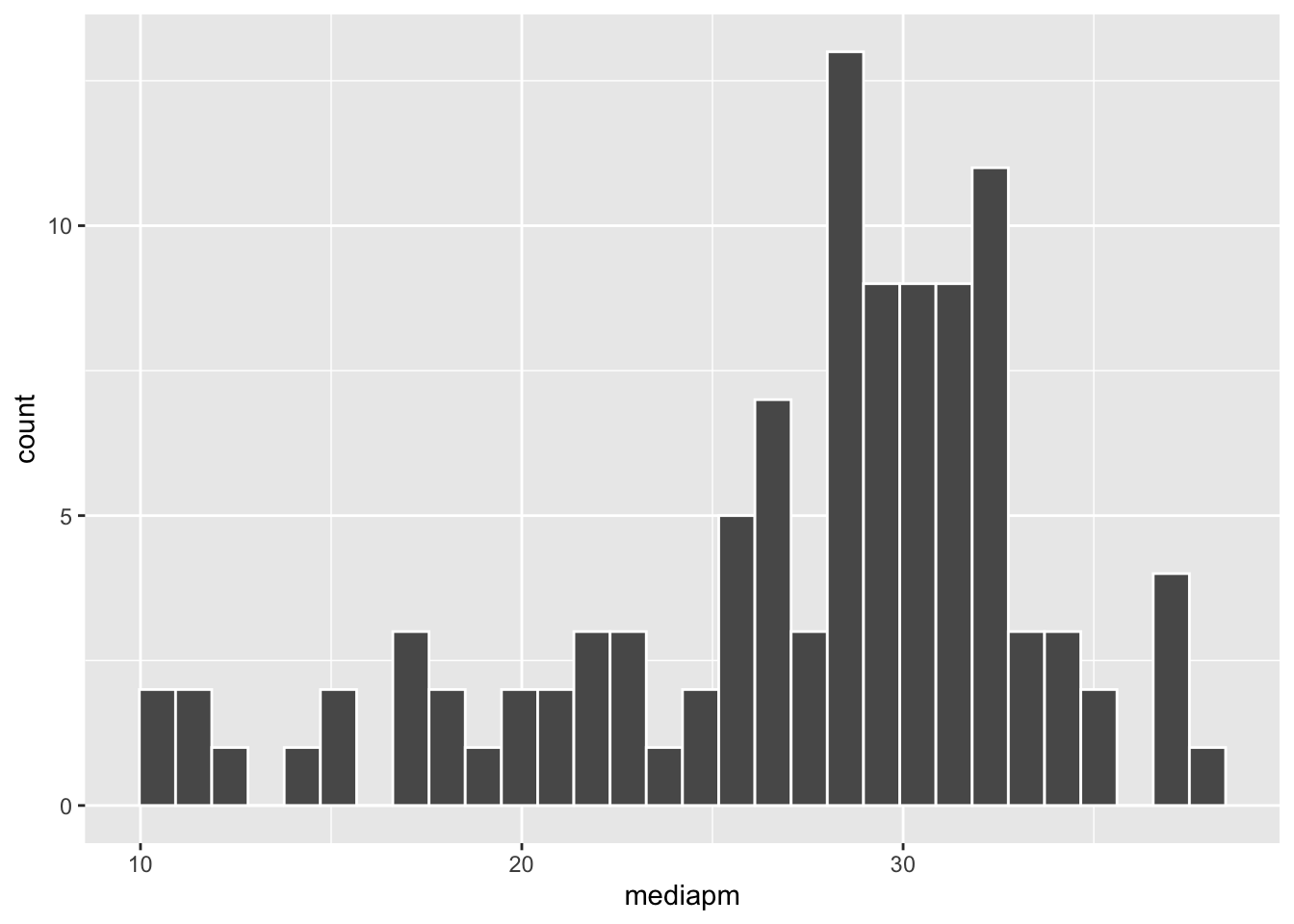
Si nota una distribuzione asimmetrica con coda lunga verso sinistra. Calcoliamo quindi qualche statistica descrittiva per la variabile mediapm:
ariasf %>%
summarise(min(mediapm),
mean(mediapm),
median(mediapm),
var(mediapm))Simple feature collection with 1 feature and 4 fields
Geometry type: MULTIPOINT
Dimension: XY
Bounding box: xmin: 444233.8 ymin: 4935495 xmax: 709473.9 ymax: 5164557
Projected CRS: WGS 84 / UTM zone 32N
# A tibble: 1 × 5
`min(mediapm)` `mean(mediapm)` `median(mediapm)` `var(mediapm)`
<dbl> <dbl> <dbl> <dbl>
1 10.1 27.3 28.7 39.6
# ℹ 1 more variable: geometry <MULTIPOINT [m]>Rappresentiamo ora graficamente con una mappa la distribuzione di mediapm (ogni punto corrisponde ad una stazione di monitoraggio):
library(sf)
ariasf %>%
ggplot() +
geom_sf(aes(col=mediapm))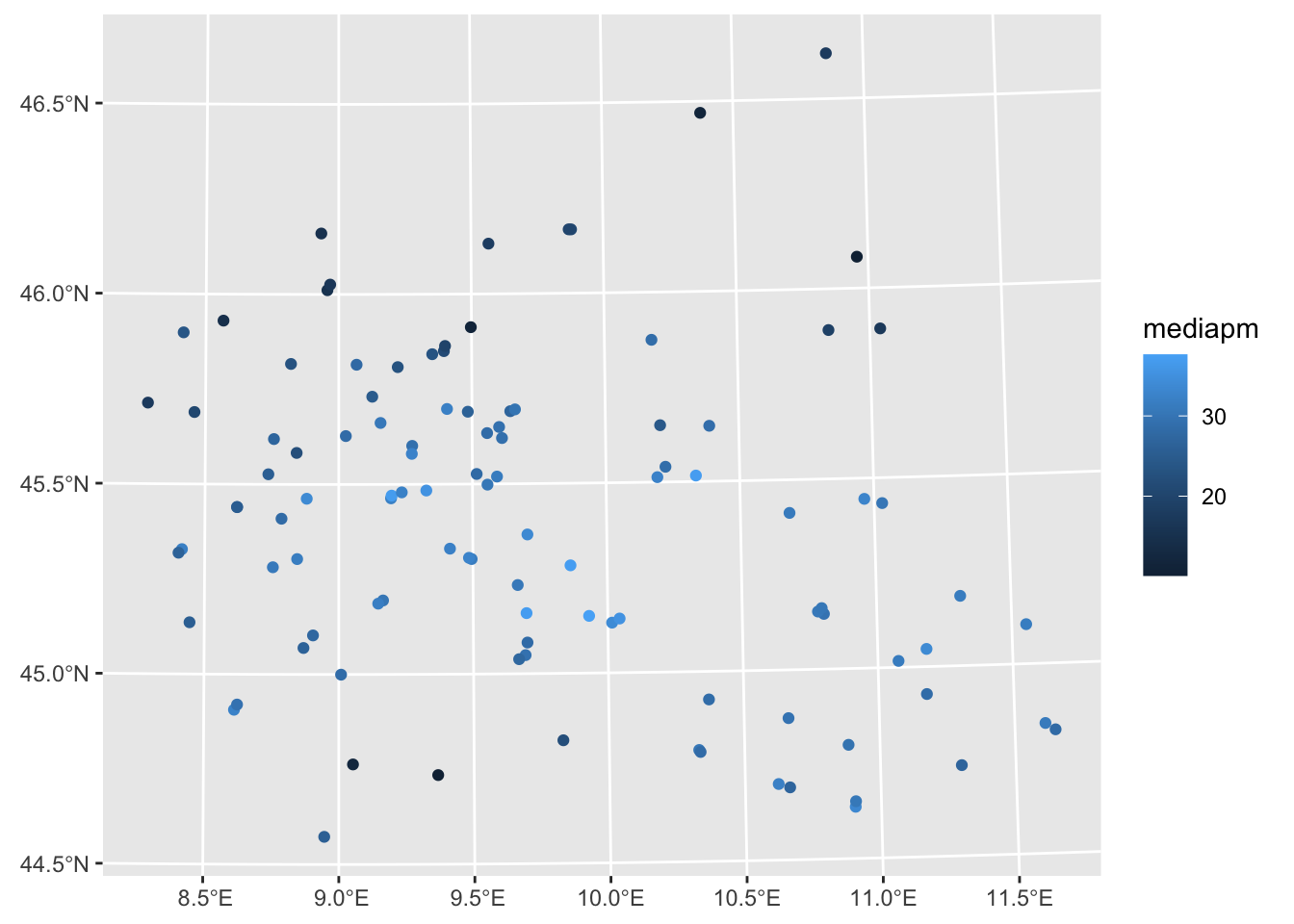
Usando la scala di colori di default, valori chiari corrispondono a concentrazioni medie annue maggiori.
Andiamo ora prevedere la concentrazione (media annua) di PM10 su tutta l’area di interesse usando il metodo della distanza inversa ponderata (si vedano anche le slide MAD2324_Lez9.pdf). Di fatto la previsione in una nuova localizzazione è la media pesata dei valori osservati di PM10 nelle centraline. I pesi sono inversamente proporzionali alla distanza tra la nuova localizzazione e le centraline (ovviamente centraline più vicine avranno un’importanza maggiore).
Procediamo come segue:
griglia = st_make_grid(ariasf, n=c(10,10))Rappresentiamo graficamente la griglia (griglia) insieme alle stazioni di monitoraggio (i cui dati sono nel file ariasf):
griglia %>%
ggplot() +
geom_sf() +
geom_sf(aes(col=mediapm), data = ariasf)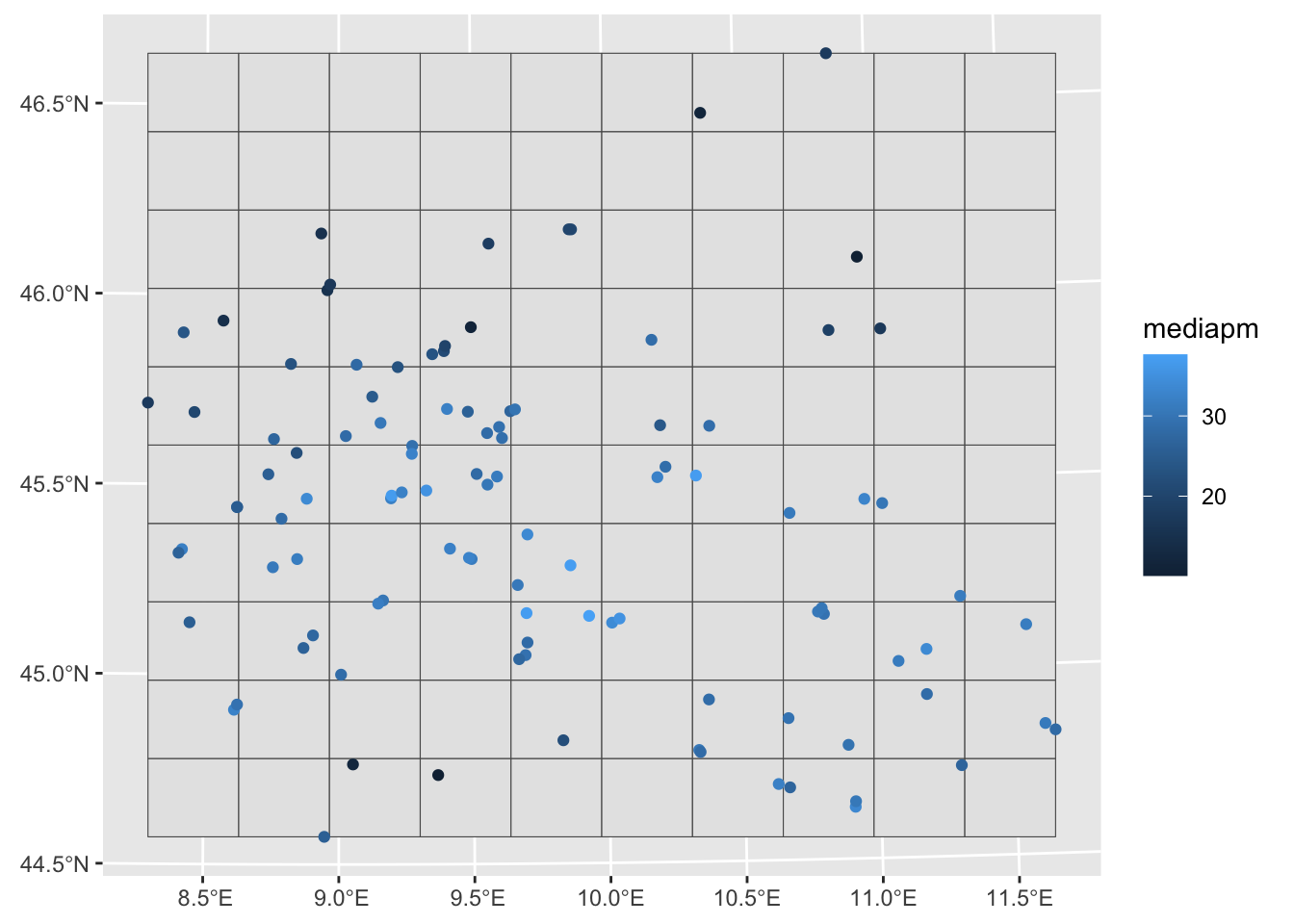
idw contenuta nel pacchetto gstat:# library(gstat)
interp = idw(mediapm ~ 1, # variabile da prevedere
ariasf, # dati relativi alle stazioni di monitoraggio
griglia) # nuove localizzazioni spaziali[inverse distance weighted interpolation]head(interp)Simple feature collection with 6 features and 4 fields
Geometry type: POLYGON
Dimension: XY
Bounding box: xmin: 444233.8 ymin: 4935495 xmax: 603377.9 ymax: 4958401
Projected CRS: WGS 84 / UTM zone 32N
x y var1.pred var1.var geometry
ID1 457495.8 4946948 27.72914 NA POLYGON ((444233.8 4935495,...
ID2 484019.8 4946948 25.80336 NA POLYGON ((470757.8 4935495,...
ID3 510543.9 4946948 20.97625 NA POLYGON ((497281.9 4935495,...
ID4 537067.9 4946948 21.23865 NA POLYGON ((523805.9 4935495,...
ID5 563591.9 4946948 26.87564 NA POLYGON ((550329.9 4935495,...
ID6 590115.9 4946948 28.78700 NA POLYGON ((576853.9 4935495,...Si noti che il nuovo oggetto interp contiene diverse colonne. Quella per noi importante è var1.pred ovvero quella che contiene le previsioni.
var1.pred insieme ai valori delle centraline:interp %>%
ggplot()+
geom_sf(aes(fill = var1.pred)) +
geom_sf(aes(col=mediapm), data = ariasf) +
scale_fill_gradient(low="yellow", high="red")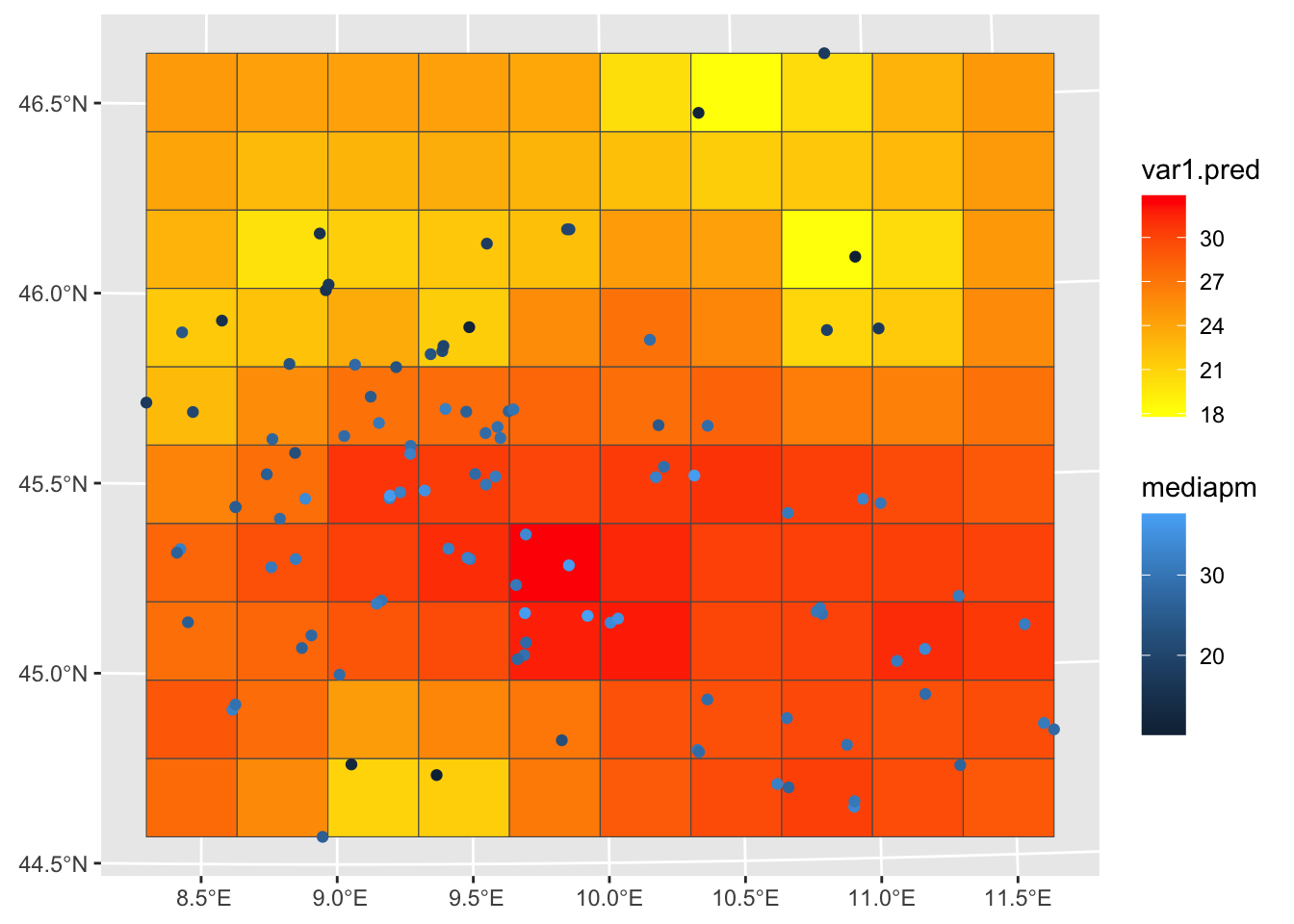
Per i valori sulla griglia utilizziamo una scala di colori diversa (si noti la presenza di due legende).
E’ possibile ripetere l’analisi usando una griglia più fitta con, ad esempio 20 righe e 20 colonne (400 celle):
griglia = st_make_grid(ariasf, n=c(20,20))
interp = idw(mediapm ~ 1, # variabile da prevedere
ariasf, # dati relativi alle stazioni di monitoraggio
griglia) # nuove localizzazioni spaziali[inverse distance weighted interpolation]interp %>%
ggplot()+
geom_sf(aes(fill = var1.pred)) +
geom_sf(aes(col=mediapm), data = ariasf) +
scale_fill_gradient(low="yellow", high="red")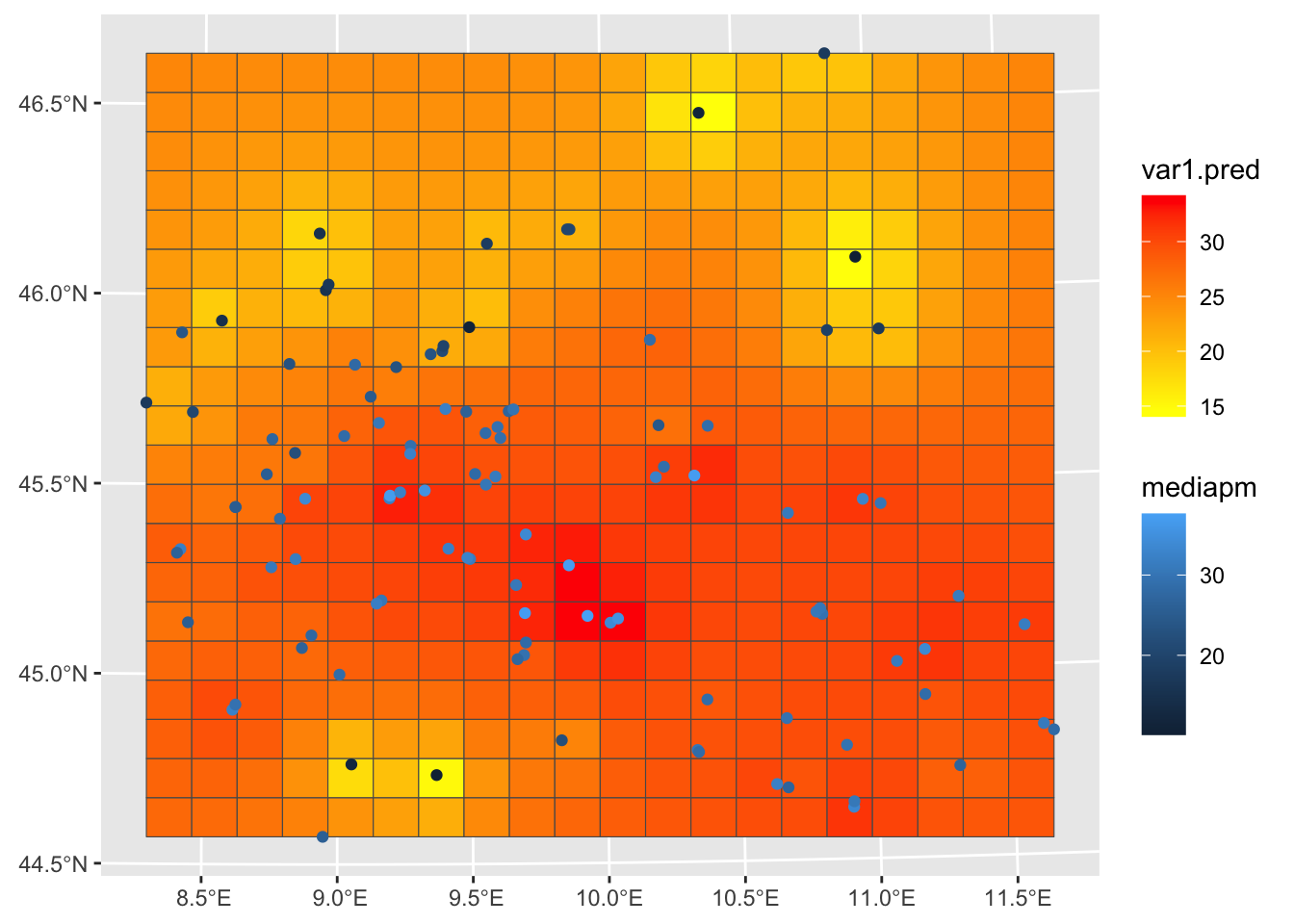
RMarkdown è un ambiente di lavoro per progetti di data science (https://rmarkdown.rstudio.com/). Esso permette di ottenere report riproducibili che combinano testo, codice di R ed il relativo output. Per un introduzione a RMarkdown si veda https://rmarkdown.rstudio.com/lesson-1.html.
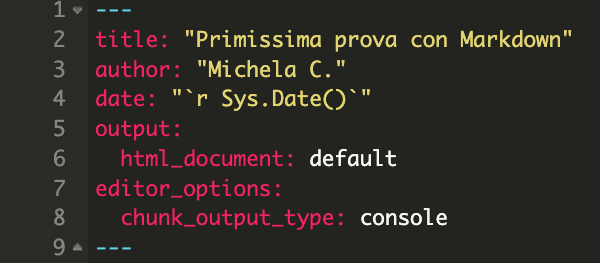
Un file RMarkdown ha estensione .rmd. Si scarichi ad esempio il file Prova1.Rmd dalla cartella Moodle “Script di R scritti in aula”. Il file può essere aperto con RStudio. Nella parte in alto del file, come mostrato in Figure 9.1, potete modificare il titolo del documento (title) e il nome dell’autore (author). Il resto (testo e simboli) non deve essere modificato. In automatico viene printata la data del giorno in cui si apre il documento.
E’ possibile compilare il file per ottenere come output un file html utilizzando il tasto con il gomitolo rappresentato dalla freccia verde in Figure 9.2). Se la compilazione si conclude correttamente, si aprirà una pagina html con il contenuto del file. Inoltre un file .html verrà automaticamente creato nella stessa cartella dove si trova il file .Rmd.
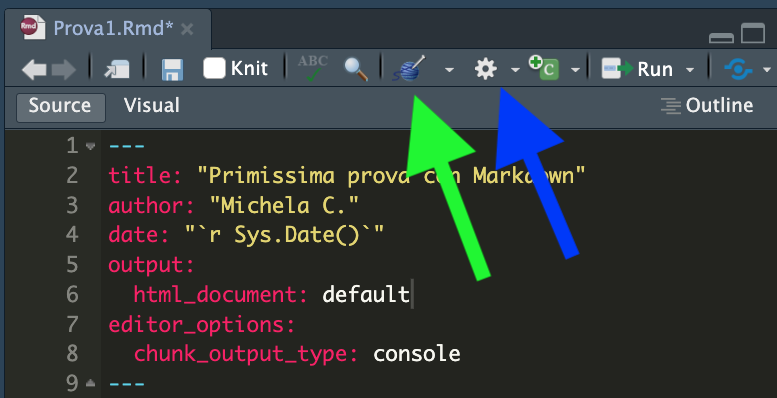
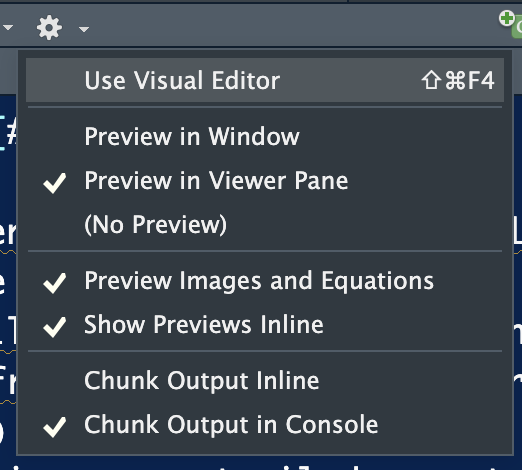
Se si vuole vedere il documento compilato nel pannello in basso a destra di Rstudio (e non nel browser, in una finestra separata) è possibile cliccare sul tasto con la rotella grigia (si veda la freccia blu in Figure 9.2 andando poi a scegliere le opzioni come mostrato in Figure 9.3) Selezionare in particolare Preview in Viewer Pane e Chunck output in Console. Si compili poi nuovamente il documento con il tasto gomitolo, il documento finale verrà poi mostrato nella scheda Viewer pane in basso a destra.
In Figure 9.4 è possibile vedere una parte di file RMarkdown. Nella parte superiore si vede del semplice testo (l’utilizzo delle ... permette di utilizzare un font speciale che richiama quello di R). La parte con sfondo più chiaro (grigio chiaro) è detta “Executable cell” e contiene codice R che poi verrà eseguito quando si compila il file (facendo vedere i relativi output).
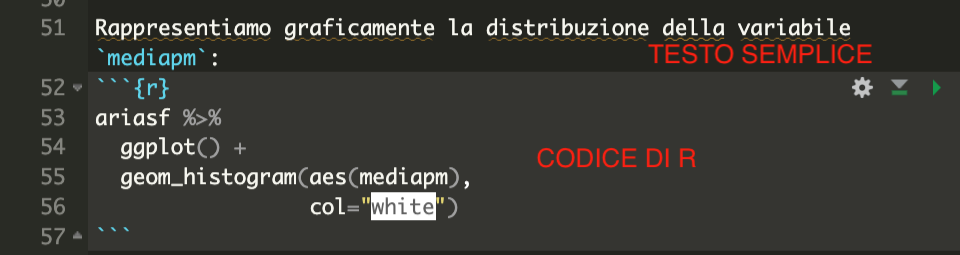
E’ anche possibile controllare cosa il proprio codice produce in output facendo girare separatamente ogni singola linea del codice utilizzando i tasti Ctrl/Cmd Enter. Questo è di fatto l’approccio che è stato utilizzato fino ad ora per far girare del codice e vedere i corrispondenti risultati nella Console. I risultati verranno mostrati nella console e i nuovi oggetti salvati nell’ambiente di lavoro (si veda il pannello in alto a destra). E’ necessario in ogni caso compilare l’intero documento con il tasto gomitolo se si vuole vedere il report finale aggiornato nella sua versione html.