library(sf)Linking to GEOS 3.11.0, GDAL 3.5.3, PROJ 9.1.0; sf_use_s2() is TRUElibrary(mapview)In questa lezione vedremo:
come costruire una mappa tematica usando lo shapefile delle regioni italiane,
come rappresentare in una mappa dei punti di cui si conoscono le coordinate geografiche.
Saranno necessari due pacchetti: sf e mapview da installare, secondo quanto descritto in Section 4.2, e poi caricare:
library(sf)Linking to GEOS 3.11.0, GDAL 3.5.3, PROJ 9.1.0; sf_use_s2() is TRUElibrary(mapview)Il pacchetto sf è corredato da un libro on-line dal titolo Spatial Data Science with applications in R disponibile al seguente link: https://r-spatial.org/book/
Carichiamo anche la solita libreria tidyverse:
library(tidyverse)── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.4
✔ forcats 1.0.0 ✔ stringr 1.5.0
✔ ggplot2 3.5.1 ✔ tibble 3.2.1
✔ lubridate 1.9.2 ✔ tidyr 1.3.0
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorsPer questo laboratorio verranno utilizzati due file csv (disponibili nella cartella Dati della pagina Moodle del corso):
Ripartizione: ripartizione geografica in cui si trova la regionecodreg: codice regionedenreg: nome regione2014: densità di piste ciclabili per il 20142021: densità di piste ciclabili per il 2021Verranno inoltre utilizzati due shapefile i cui file sono disponibili nella cartella ShapefilexLabCartella della pagina Moodle del corso:
Per importare uno shapefile si utilizza la funzione st_read che prevede di specificare il percorso (tra virgolette) del file *.shp nel proprio computer. Per recupeare il percorso si può utilizzare la funzione file.choose() che, dopo aver selezionato il file di interesse, ne restituisce il percorso come stringa di testo (che andrà poi copiata).
reg = st_read("./data/Reg01012023_g_WGS84.shp")Reading layer `Reg01012023_g_WGS84' from data source
`/Users/michelacameletti/Library/CloudStorage/GoogleDrive-michela.cameletti@unibg.it/My Drive/UniBg/Didattica/Lingue_GeoUrb/2023-2024/MAD2324Rlabs/data/Reg01012023_g_WGS84.shp'
using driver `ESRI Shapefile'
Simple feature collection with 20 features and 5 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 313279.3 ymin: 3933846 xmax: 1312016 ymax: 5220292
Projected CRS: WGS 84 / UTM zone 32Nclass(reg)[1] "sf" "data.frame"L’oggetto reg è un data frame che ha in aggiunta una geometria (in questo caso le informazioni relative ai poligoni) come si vede nella struttura di reg:
glimpse(reg)Rows: 20
Columns: 6
$ COD_RIP <dbl> 1, 1, 1, 2, 2, 2, 1, 2, 3, 3, 3, 3, 4, 4, 4, 4, 4, 4, 5, 5
$ COD_REG <dbl> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, …
$ DEN_REG <chr> "Piemonte", "Valle d'Aosta", "Lombardia", "Trentino-Alto Ad…
$ Shape_Leng <dbl> 1236799.8, 310968.1, 1410222.9, 800893.7, 1054587.0, 670820…
$ Shape_Area <dbl> 25393881930, 3258837561, 23862315752, 13607548167, 18343552…
$ geometry <MULTIPOLYGON [m]> MULTIPOLYGON (((457749.5 51..., MULTIPOLYGON (((390652.6 50…Essendo un data frame si possono utilizzare i verbi di tidyverse imparati nei precedenti laboratori. E’ possibile, ad esempio, usare filter per selezionare una regione:
reg %>%
filter(DEN_REG == "Lombardia")Simple feature collection with 1 feature and 5 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 460662.8 ymin: 4947430 xmax: 691489.7 ymax: 5165371
Projected CRS: WGS 84 / UTM zone 32N
COD_RIP COD_REG DEN_REG Shape_Leng Shape_Area
1 1 3 Lombardia 1410223 23862315752
geometry
1 MULTIPOLYGON (((485536.4 49...La funzione st_crs restituisce tutte le informazioni inerenti il sistema di riferimento delle coordinate per reg (si noti che per questo shapefile le coordinate sono di tipo UTMX-UTMY):
st_crs(reg)Coordinate Reference System:
User input: WGS 84 / UTM zone 32N
wkt:
PROJCRS["WGS 84 / UTM zone 32N",
BASEGEOGCRS["WGS 84",
DATUM["World Geodetic System 1984",
ELLIPSOID["WGS 84",6378137,298.257223563,
LENGTHUNIT["metre",1]]],
PRIMEM["Greenwich",0,
ANGLEUNIT["degree",0.0174532925199433]],
ID["EPSG",4326]],
CONVERSION["UTM zone 32N",
METHOD["Transverse Mercator",
ID["EPSG",9807]],
PARAMETER["Latitude of natural origin",0,
ANGLEUNIT["Degree",0.0174532925199433],
ID["EPSG",8801]],
PARAMETER["Longitude of natural origin",9,
ANGLEUNIT["Degree",0.0174532925199433],
ID["EPSG",8802]],
PARAMETER["Scale factor at natural origin",0.9996,
SCALEUNIT["unity",1],
ID["EPSG",8805]],
PARAMETER["False easting",500000,
LENGTHUNIT["metre",1],
ID["EPSG",8806]],
PARAMETER["False northing",0,
LENGTHUNIT["metre",1],
ID["EPSG",8807]]],
CS[Cartesian,2],
AXIS["(E)",east,
ORDER[1],
LENGTHUNIT["metre",1]],
AXIS["(N)",north,
ORDER[2],
LENGTHUNIT["metre",1]],
ID["EPSG",32632]]Per rappresentare la geometria di reg si utilizza ggplot con geometria geom_sf:
reg %>%
ggplot() +
geom_sf()
E’ possibile cambiare il bordo delle regioni e il colore di riempimento come segue:
reg %>%
ggplot() +
geom_sf(col="blue",
fill="lightgreen") #colore uguale per tutte le regioni
E’ più interessante utilizzare, come colore di riempimento, i valori di una variabile disponibile in reg come, ad esempio, Shape_Area. In questo caso fill andrà specificato dentro aes (essendo definito da una variabile e non da un colore fisso):
reg %>%
ggplot() +
geom_sf(aes(fill=Shape_Area), col="blue")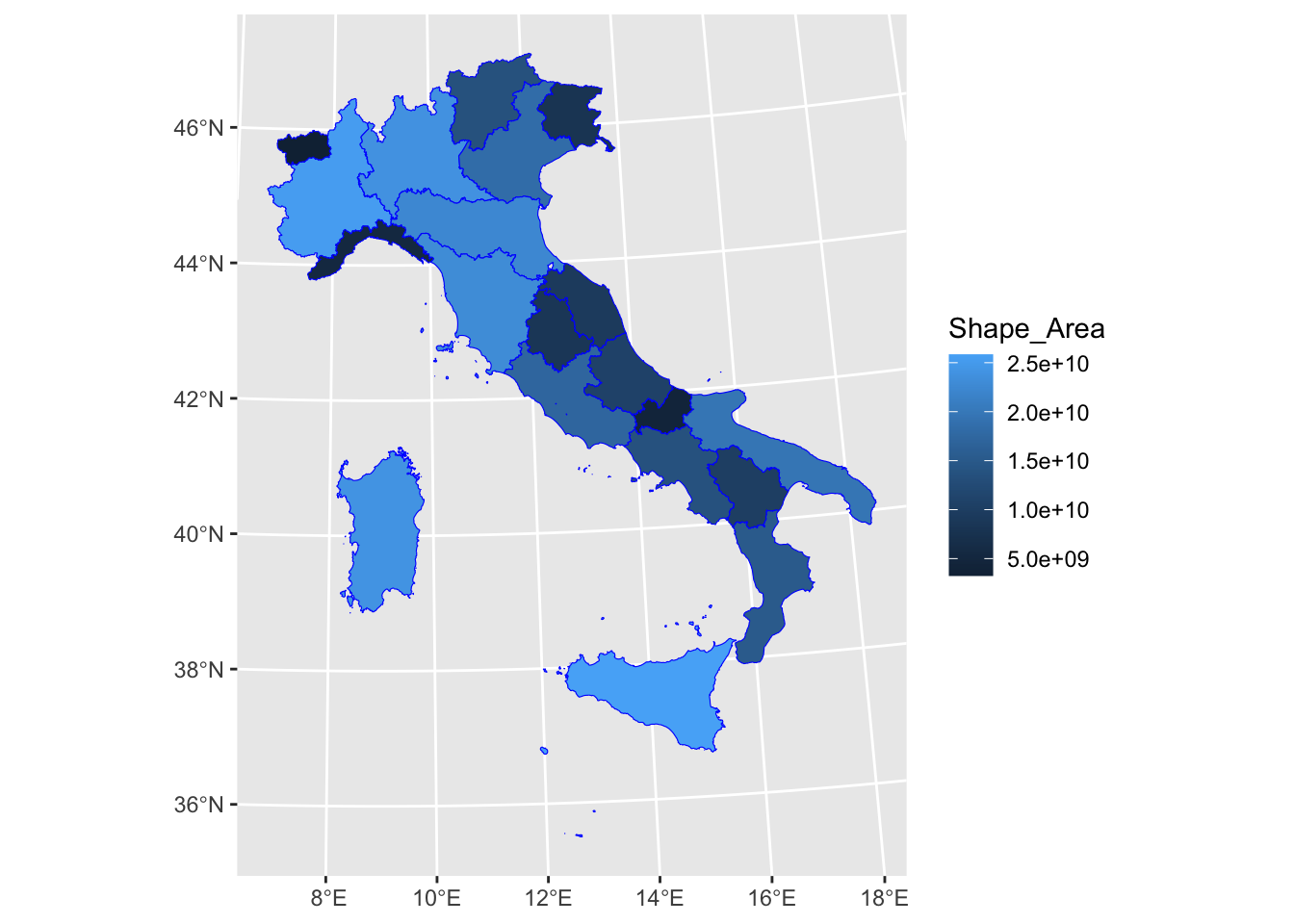
La scala continua di colori è definita in automatico dal blu scuro (valori bassi) a blu chiaro (valori alti di densità di piste ciclabili). E’ possibile cambiare la scala di colori andando per esempio ad utilizzare un gradiente giallo-rosso:
reg %>%
ggplot() +
geom_sf(aes(fill=Shape_Area), col="blue") +
scale_fill_gradient(low="yellow", high="red")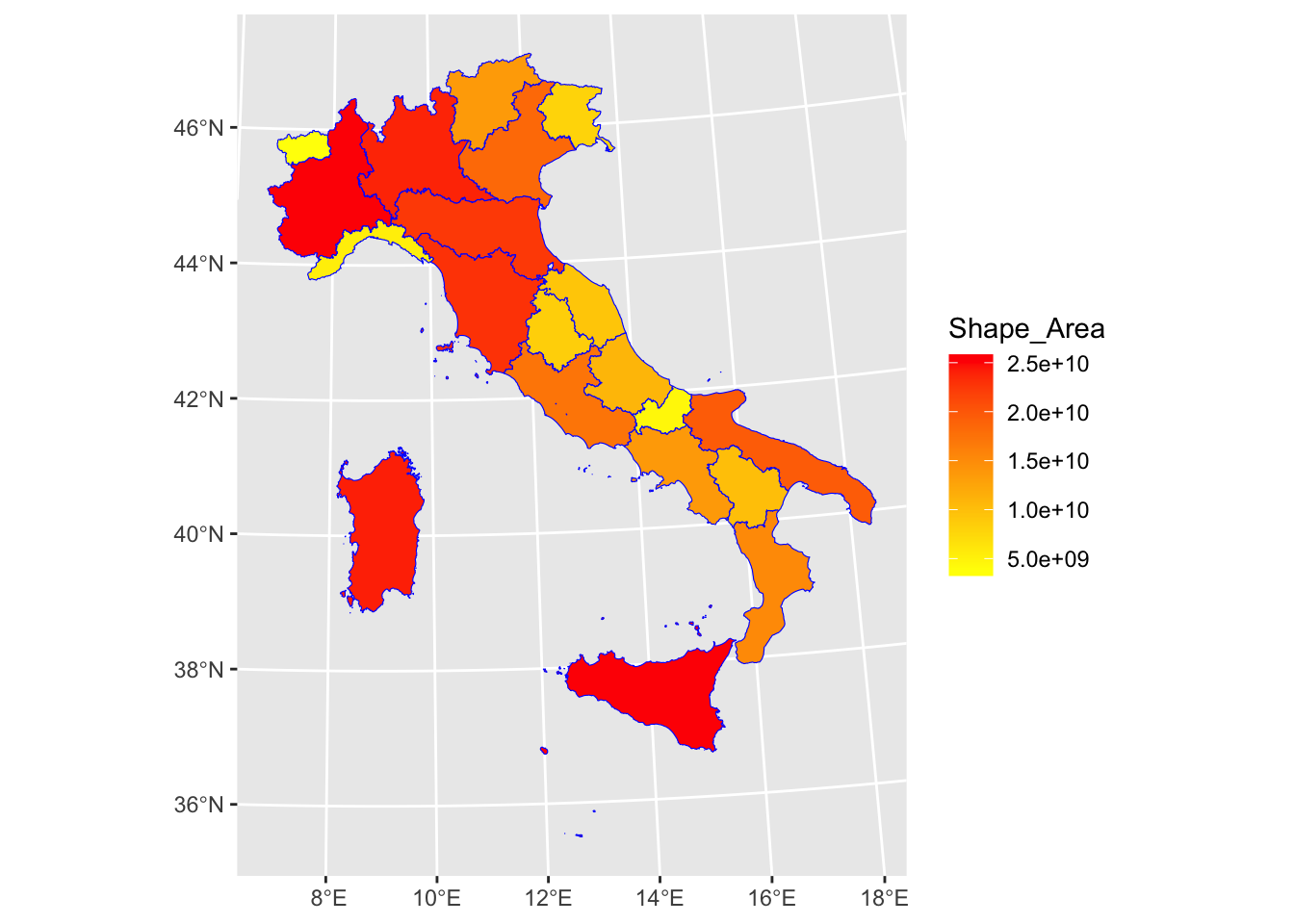
Per maggiori informazioni sulle scale continue di colori si veda https://ggplot2.tidyverse.org/reference/scale_colour_continuous.html.
Importiamo ora il file bici.csv:
bici <- read.csv("./data/bici.csv", stringsAsFactors = T)
glimpse(bici)Rows: 20
Columns: 11
$ Ripartizione <fct> Nord-ovest, Nord-ovest, Nord-ovest, Nord-est, Nord-est, N…
$ codreg <int> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17…
$ denreg <fct> Piemonte, Valle d'Aosta/Vallée d'Aoste, Lombardia, Trenti…
$ X2014 <dbl> 38.2, 34.6, 85.5, 50.7, 47.0, 41.0, 4.4, 51.6, 17.9, 4.1,…
$ X2015 <dbl> 40.3, 34.6, 90.6, 52.9, 47.9, 41.2, 4.8, 52.9, 18.2, 4.1,…
$ X2016 <dbl> 41.8, 34.6, 95.3, 54.2, 49.0, 44.8, 4.9, 54.6, 18.3, 4.1,…
$ X2017 <dbl> 43.2, 34.6, 98.5, 56.2, 49.2, 43.0, 7.0, 56.3, 19.0, 4.1,…
$ X2018 <dbl> 44.5, 34.6, 102.0, 56.6, 51.4, 44.1, 7.0, 57.4, 19.2, 4.9…
$ X2019 <dbl> 46.2, 34.6, 106.5, 57.4, 53.1, 45.3, 7.5, 59.2, 23.0, 5.2…
$ X2020 <dbl> 44.8, 35.0, 118.4, 59.4, 55.8, 57.5, 12.0, 60.9, 21.4, 5.…
$ X2021 <dbl> 49.7, 34.6, 122.8, 66.9, 58.2, 59.7, 18.9, 64.2, 24.1, 6.…Si noti che in automatico R ha aggiunto X davanti ai nomi delle colonne 2014, …, 2021 perchè non è possibile avere nomi di colonne che iniziano con un numero.
Procediamo ora ad unire lo shapefile delle regioni italiane (reg) e il file relativo alle piste ciclabili (bici). L’unione viene fatta utilizzando come variabile chiave il codice regione che è dato dalla variabile COD_REG nello shapefile e codreg nell’oggetto bici. L’unione si realizza con la funzione inner_join (per maggiori dettagli su tutte le operazioni di join vedere ad esempio https://mikoontz.github.io/data-carpentry-week/lesson_joins.html). Tramite by e join_by andiamo a specificare la variabile chiave:
reg2 = reg %>%
inner_join(bici,
by = join_by(COD_REG == codreg))
glimpse(reg2)Rows: 20
Columns: 16
$ COD_RIP <dbl> 1, 1, 1, 2, 2, 2, 1, 2, 3, 3, 3, 3, 4, 4, 4, 4, 4, 4, 5, 5
$ COD_REG <dbl> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17…
$ DEN_REG <chr> "Piemonte", "Valle d'Aosta", "Lombardia", "Trentino-Alto …
$ Shape_Leng <dbl> 1236799.8, 310968.1, 1410222.9, 800893.7, 1054587.0, 6708…
$ Shape_Area <dbl> 25393881930, 3258837561, 23862315752, 13607548167, 183435…
$ Ripartizione <fct> Nord-ovest, Nord-ovest, Nord-ovest, Nord-est, Nord-est, N…
$ denreg <fct> Piemonte, Valle d'Aosta/Vallée d'Aoste, Lombardia, Trenti…
$ X2014 <dbl> 38.2, 34.6, 85.5, 50.7, 47.0, 41.0, 4.4, 51.6, 17.9, 4.1,…
$ X2015 <dbl> 40.3, 34.6, 90.6, 52.9, 47.9, 41.2, 4.8, 52.9, 18.2, 4.1,…
$ X2016 <dbl> 41.8, 34.6, 95.3, 54.2, 49.0, 44.8, 4.9, 54.6, 18.3, 4.1,…
$ X2017 <dbl> 43.2, 34.6, 98.5, 56.2, 49.2, 43.0, 7.0, 56.3, 19.0, 4.1,…
$ X2018 <dbl> 44.5, 34.6, 102.0, 56.6, 51.4, 44.1, 7.0, 57.4, 19.2, 4.9…
$ X2019 <dbl> 46.2, 34.6, 106.5, 57.4, 53.1, 45.3, 7.5, 59.2, 23.0, 5.2…
$ X2020 <dbl> 44.8, 35.0, 118.4, 59.4, 55.8, 57.5, 12.0, 60.9, 21.4, 5.…
$ X2021 <dbl> 49.7, 34.6, 122.8, 66.9, 58.2, 59.7, 18.9, 64.2, 24.1, 6.…
$ geometry <MULTIPOLYGON [m]> MULTIPOLYGON (((457749.5 51..., MULTIPOLYGON (((390652.6 …La tabella degli attributi dello shapefile reg2 contiene ora le variabili relative alle piste ciclabili. Facciamo ora la rappresentazione tematica di una di esse (X2014):
reg2 %>%
ggplot() +
geom_sf(aes(fill=X2014), col="black") +
scale_fill_gradient(low="yellow", high="red") +
ggtitle("Densità di piste ciclabili - anno 2014")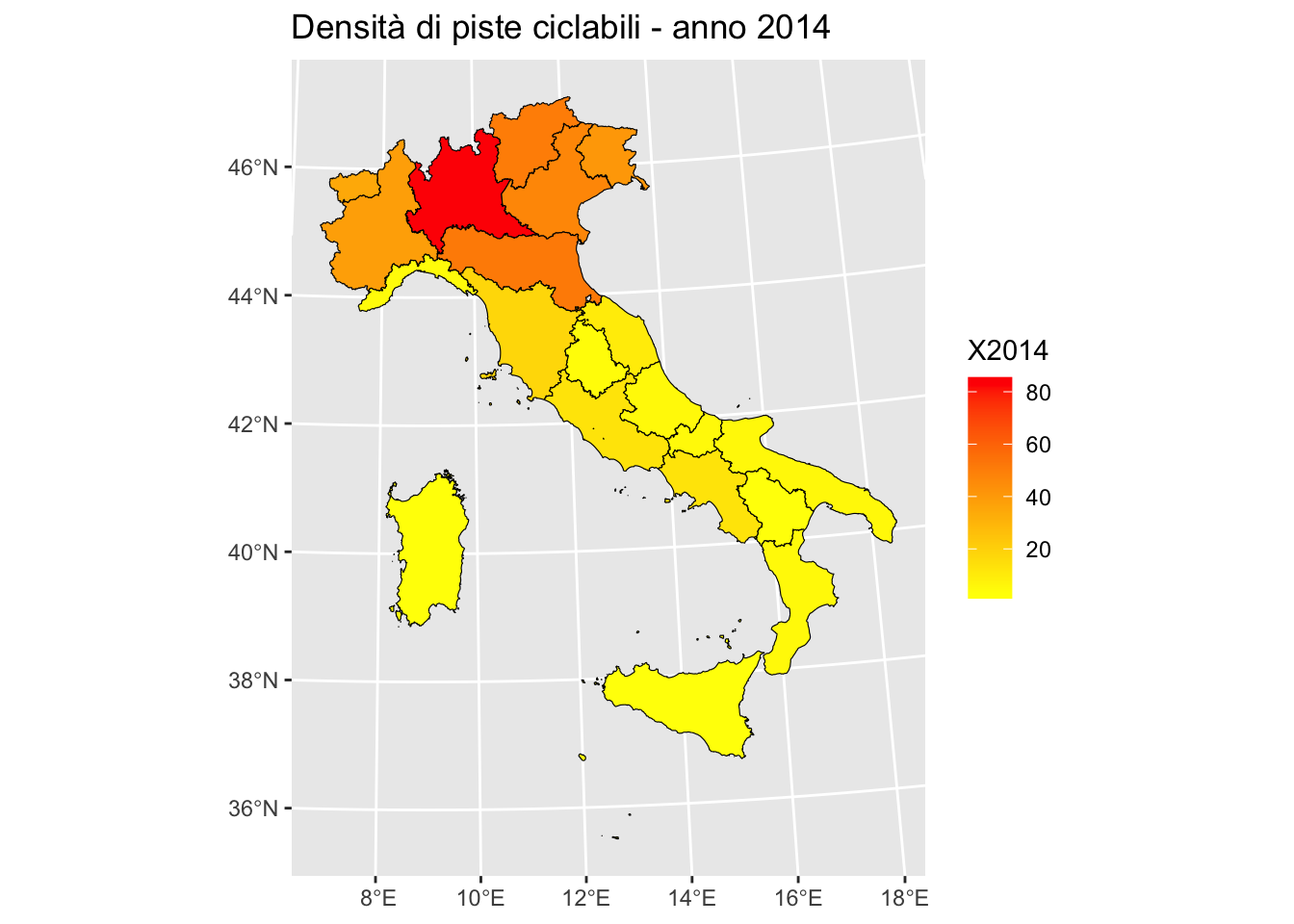
Sarebbe interessante avere una mappa per ogni anno senza dover ripetere il codice sopra riportato per ogni singolo anno. A tal fine è necessario ridimensionare il file da wide a long come descritto in Section 6.3:
reg2long = reg2 %>%
pivot_longer(X2014:X2021,
names_to = "anno",
values_to = "kmpiste")
glimpse(reg2long)Rows: 160
Columns: 10
$ COD_RIP <dbl> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, …
$ COD_REG <dbl> 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 3, 3, 3, …
$ DEN_REG <chr> "Piemonte", "Piemonte", "Piemonte", "Piemonte", "Piemonte…
$ Shape_Leng <dbl> 1236799.8, 1236799.8, 1236799.8, 1236799.8, 1236799.8, 12…
$ Shape_Area <dbl> 25393881930, 25393881930, 25393881930, 25393881930, 25393…
$ Ripartizione <fct> Nord-ovest, Nord-ovest, Nord-ovest, Nord-ovest, Nord-oves…
$ denreg <fct> Piemonte, Piemonte, Piemonte, Piemonte, Piemonte, Piemont…
$ geometry <MULTIPOLYGON [m]> MULTIPOLYGON (((457749.5 51..., MULTIPOLYGON…
$ anno <chr> "X2014", "X2015", "X2016", "X2017", "X2018", "X2019", "X2…
$ kmpiste <dbl> 38.2, 40.3, 41.8, 43.2, 44.5, 46.2, 44.8, 49.7, 34.6, 34.…Abbiamo ora due variabili anno e kmpiste che verranno utilizzate per ottenere le 8 mappe. Prima di procedere eliminiamo le X che sono presenti nella colonna anno:
reg2long %>% count(anno)Simple feature collection with 8 features and 2 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 313279.3 ymin: 3933846 xmax: 1312016 ymax: 5220292
Projected CRS: WGS 84 / UTM zone 32N
# A tibble: 8 × 3
anno n geometry
* <chr> <int> <MULTIPOLYGON [m]>
1 X2014 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
2 X2015 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
3 X2016 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
4 X2017 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
5 X2018 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
6 X2019 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
7 X2020 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…
8 X2021 20 (((449018.8 4301546, 448996.8 4301476, 448980.8 4301447, 448886.9…Come descritto in Section 6.3 a tale scopo viene utilizzate la funzione str_replace:
reg2long = reg2long %>%
mutate(anno = str_replace(anno,"X",""))Procediamo ora con la rappresentazione grafica utilizzando kmpiste per il colore di riempimento e anno per facet_wrap (per creare una mappa separata per ogni anno):
reg2long %>%
ggplot()+
geom_sf(aes(fill=kmpiste)) +
facet_wrap(~anno) +
scale_fill_gradient(low="yellow", high="red") 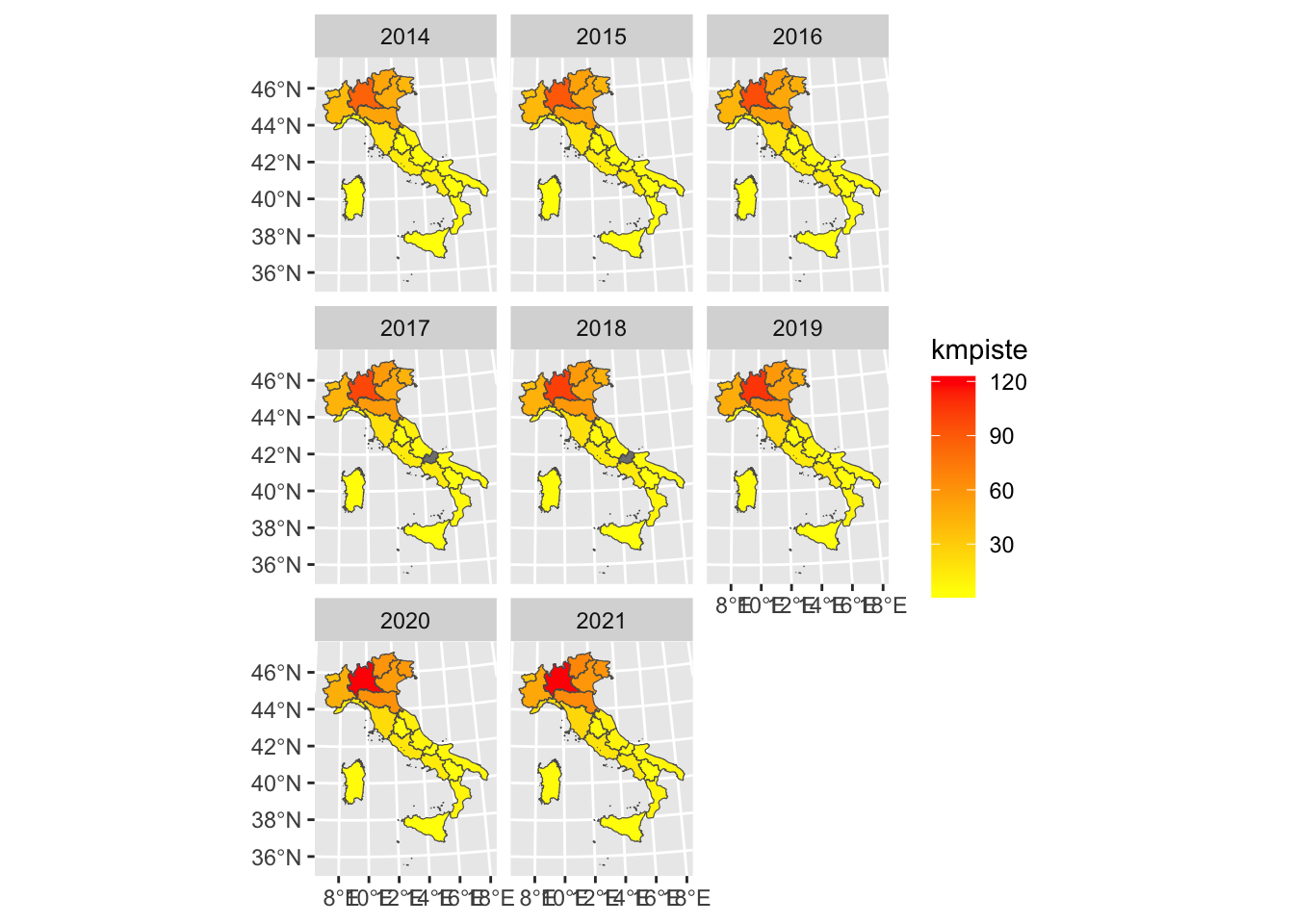
Si nota che nelle regioni del nord la densità di piste ciclabili è maggiore. Si nota anche un miglioramento nel corso degli anni. Per semplicità è possibile selezionare solo il primo e ultimo anno:
reg2long %>%
filter(anno==2014 | anno==2021) %>%
ggplot()+
geom_sf(aes(fill=kmpiste)) +
facet_wrap(~anno) +
scale_fill_gradient(low="yellow", high="red") 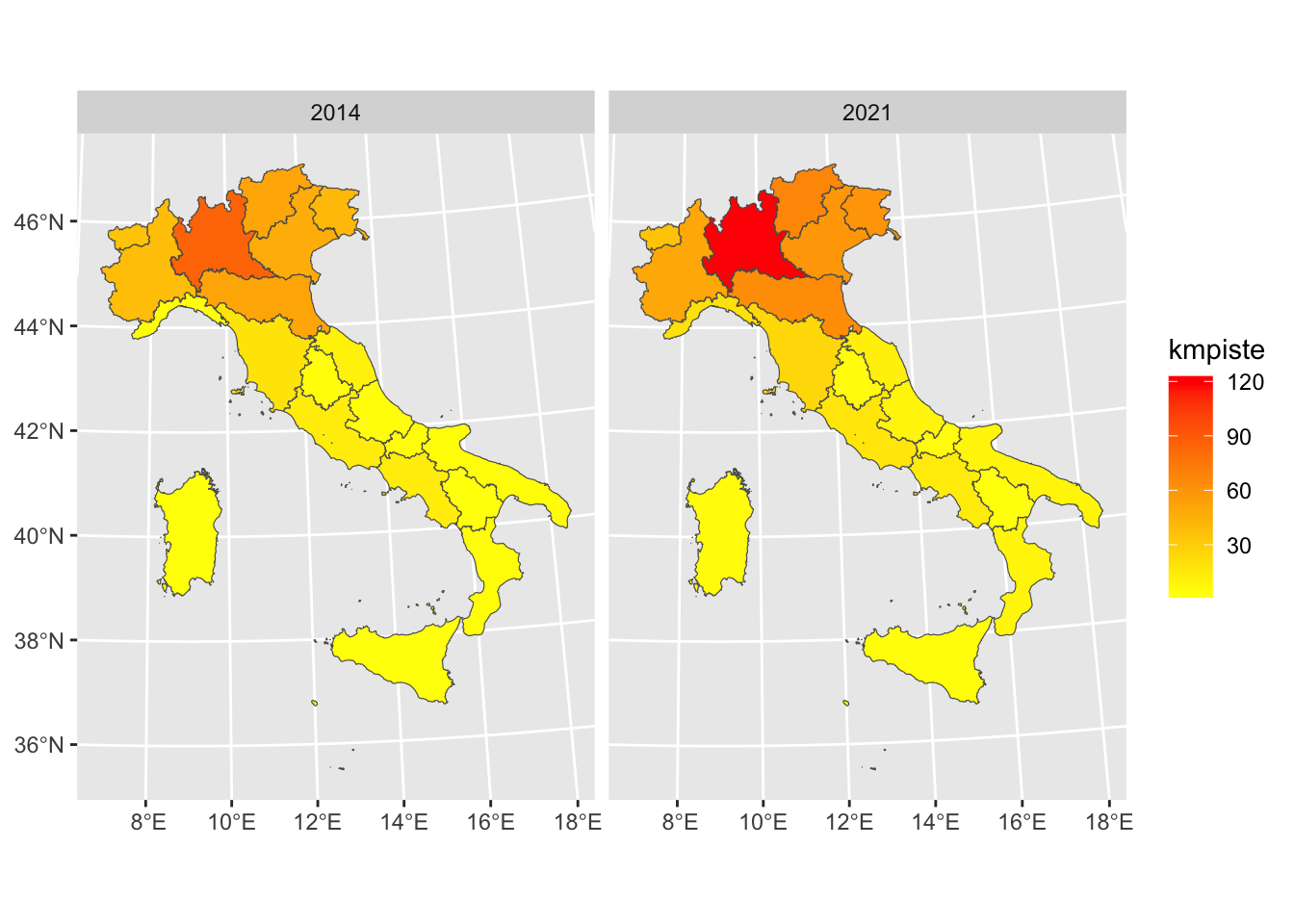
Nel verbo filter si usa l’operatore | che rappresenta l’operazione di unione (anno 2014 oppure anno 2021).
Quanto visto sopra permette di ottenere mappe statistiche. Se si è interessati ad ottenere una mappa interattiva per un singolo anno è possibile utilizzare la funzione mapview. Tornando a reg2 si consideri ad esempio X2014:
reg2 %>%
mapview(zcol = "X2014")reg2 %>%
mapview(zcol = c("X2014","X2015"))Per maggiori dettagli si veda https://r-spatial.github.io/mapview/.
Importiamo il file Agrimonia_Dataset_v_3_0_0.csv andando a creare un oggetto di nome aria:
aria <- read.csv("./data/Agrimonia_Dataset_v_3_0_0.csv",
stringsAsFactors = T)
glimpse(aria)Rows: 309,072
Columns: 43
$ IDStations <fct> 1264, 1264, 1264, 1264, 1264, 1264, 1264, …
$ Latitude <dbl> 46.16785, 46.16785, 46.16785, 46.16785, 46…
$ Longitude <dbl> 9.87921, 9.87921, 9.87921, 9.87921, 9.8792…
$ Time <fct> 2016-01-01, 2016-01-02, 2016-01-03, 2016-0…
$ Altitude <int> 290, 290, 290, 290, 290, 290, 290, 290, 29…
$ AQ_pm10 <dbl> 62, 73, 44, 31, 27, 27, 43, 37, 48, NaN, N…
$ AQ_pm25 <dbl> 53, 63, 39, 29, 26, 25, 38, 32, 45, NaN, N…
$ AQ_co <dbl> NaN, NaN, NaN, NaN, NaN, NaN, NaN, NaN, Na…
$ AQ_nh3 <dbl> NaN, NaN, NaN, NaN, NaN, NaN, NaN, NaN, Na…
$ AQ_nox <dbl> 82.63, 101.70, 66.26, 63.31, 75.53, 61.36,…
$ AQ_no2 <dbl> 37.72, 38.84, 32.34, 31.67, 33.33, 34.40, …
$ AQ_so2 <dbl> NaN, NaN, NaN, NaN, NaN, NaN, NaN, NaN, Na…
$ WE_temp_2m <dbl> -2.2120, -3.0630, -2.7680, -4.3520, -6.184…
$ WE_wind_speed_10m_mean <dbl> 0.5970, 0.4402, 0.6954, 0.5812, 0.5906, 0.…
$ WE_wind_speed_10m_max <dbl> 0.9883, 0.7365, 1.0100, 0.9421, 0.9847, 1.…
$ WE_mode_wind_direction_10m <fct> S, SE, S, W, SE, S, S, S, SE, SE, NE, S, S…
$ WE_tot_precipitation <dbl> 2.117e-05, 8.282e-03, 2.486e-03, 4.133e-03…
$ WE_precipitation_t <int> 0, 5, 5, 5, 5, 0, 0, 0, 1, 1, 1, 5, 0, 5, …
$ WE_surface_pressure <dbl> 83720, 82990, 82320, 81410, 81530, 81660, …
$ WE_solar_radiation <dbl> 5664000, 1335000, 3728000, 1846000, 173300…
$ WE_rh_min <dbl> 58.11, 75.57, 62.20, 64.27, 58.48, 48.94, …
$ WE_rh_mean <dbl> 74.66, 89.69, 82.27, 83.39, 73.85, 64.94, …
$ WE_rh_max <dbl> 96.33, 96.61, 93.49, 97.39, 95.66, 85.71, …
$ WE_wind_speed_100m_mean <dbl> 1.0730, 1.0530, 1.9810, 1.2080, 0.8226, 1.…
$ WE_wind_speed_100m_max <dbl> 1.977, 2.321, 2.942, 2.284, 1.672, 2.379, …
$ WE_mode_wind_direction_100m <fct> S, S, S, N, SE, SE, S, S, SE, N, N, S, S, …
$ WE_blh_layer_max <dbl> 340.60, 238.70, 407.00, 536.70, 472.40, 59…
$ WE_blh_layer_min <dbl> 10.63, 21.55, 22.80, 12.21, 10.15, 13.31, …
$ EM_nh3_livestock_mm <dbl> 0.2015, 0.2020, 0.2024, 0.2027, 0.2031, 0.…
$ EM_nh3_agr_soils <dbl> 0.1462, 0.1589, 0.1708, 0.1821, 0.1929, 0.…
$ EM_nh3_agr_waste_burn <dbl> 0.0020190, 0.0019560, 0.0018900, 0.0018220…
$ EM_nh3_sum <dbl> 0.6166, 0.6298, 0.6424, 0.6545, 0.6660, 0.…
$ EM_nox_traffic <dbl> 0.8359, 0.8372, 0.8384, 0.8396, 0.8409, 0.…
$ EM_nox_sum <dbl> 1.720, 1.720, 1.720, 1.720, 1.720, 1.721, …
$ EM_so2_sum <dbl> 0.3906, 0.3911, 0.3916, 0.3923, 0.3931, 0.…
$ LI_pigs <dbl> 0.3832, 0.3832, 0.3832, 0.3832, 0.3832, 0.…
$ LI_bovine <dbl> 5.892, 5.892, 5.892, 5.892, 5.892, 5.892, …
$ LI_pigs_v2 <fct> 4.167e-01, 4.167e-01, 4.168e-01, 4.168e-01…
$ LI_bovine_v2 <fct> 6.467e+00, 6.463e+00, 6.458e+00, 6.454e+00…
$ LA_hvi <dbl> 3.998, 3.997, 3.997, 3.997, 3.997, 3.996, …
$ LA_lvi <dbl> 1.234, 1.234, 1.234, 1.234, 1.233, 1.233, …
$ LA_land_use <int> 112, 112, 112, 112, 112, 112, 112, 112, 11…
$ LA_soil_use <int> 17, 17, 17, 17, 17, 17, 17, 17, 17, 17, 17…Il data frame ha 309072 e n ncol(aria). Si noti che la variabile Time, che si riferisce al giorno di ogni misurazione, è di tipo fattore. E’ necessario trasformarla in una variabile di tipo calendario utilizzando la funzione ymd del pacchetto lubridate (appartenente alla collezione tidyverse). Si noti che la funzione ymd è richiesta in quando ogni stringa è del tipo anno-mese-anno.
aria = aria %>%
mutate(Time = ymd(Time))
glimpse(aria)Rows: 309,072
Columns: 43
$ IDStations <fct> 1264, 1264, 1264, 1264, 1264, 1264, 1264, …
$ Latitude <dbl> 46.16785, 46.16785, 46.16785, 46.16785, 46…
$ Longitude <dbl> 9.87921, 9.87921, 9.87921, 9.87921, 9.8792…
$ Time <date> 2016-01-01, 2016-01-02, 2016-01-03, 2016-…
$ Altitude <int> 290, 290, 290, 290, 290, 290, 290, 290, 29…
$ AQ_pm10 <dbl> 62, 73, 44, 31, 27, 27, 43, 37, 48, NaN, N…
$ AQ_pm25 <dbl> 53, 63, 39, 29, 26, 25, 38, 32, 45, NaN, N…
$ AQ_co <dbl> NaN, NaN, NaN, NaN, NaN, NaN, NaN, NaN, Na…
$ AQ_nh3 <dbl> NaN, NaN, NaN, NaN, NaN, NaN, NaN, NaN, Na…
$ AQ_nox <dbl> 82.63, 101.70, 66.26, 63.31, 75.53, 61.36,…
$ AQ_no2 <dbl> 37.72, 38.84, 32.34, 31.67, 33.33, 34.40, …
$ AQ_so2 <dbl> NaN, NaN, NaN, NaN, NaN, NaN, NaN, NaN, Na…
$ WE_temp_2m <dbl> -2.2120, -3.0630, -2.7680, -4.3520, -6.184…
$ WE_wind_speed_10m_mean <dbl> 0.5970, 0.4402, 0.6954, 0.5812, 0.5906, 0.…
$ WE_wind_speed_10m_max <dbl> 0.9883, 0.7365, 1.0100, 0.9421, 0.9847, 1.…
$ WE_mode_wind_direction_10m <fct> S, SE, S, W, SE, S, S, S, SE, SE, NE, S, S…
$ WE_tot_precipitation <dbl> 2.117e-05, 8.282e-03, 2.486e-03, 4.133e-03…
$ WE_precipitation_t <int> 0, 5, 5, 5, 5, 0, 0, 0, 1, 1, 1, 5, 0, 5, …
$ WE_surface_pressure <dbl> 83720, 82990, 82320, 81410, 81530, 81660, …
$ WE_solar_radiation <dbl> 5664000, 1335000, 3728000, 1846000, 173300…
$ WE_rh_min <dbl> 58.11, 75.57, 62.20, 64.27, 58.48, 48.94, …
$ WE_rh_mean <dbl> 74.66, 89.69, 82.27, 83.39, 73.85, 64.94, …
$ WE_rh_max <dbl> 96.33, 96.61, 93.49, 97.39, 95.66, 85.71, …
$ WE_wind_speed_100m_mean <dbl> 1.0730, 1.0530, 1.9810, 1.2080, 0.8226, 1.…
$ WE_wind_speed_100m_max <dbl> 1.977, 2.321, 2.942, 2.284, 1.672, 2.379, …
$ WE_mode_wind_direction_100m <fct> S, S, S, N, SE, SE, S, S, SE, N, N, S, S, …
$ WE_blh_layer_max <dbl> 340.60, 238.70, 407.00, 536.70, 472.40, 59…
$ WE_blh_layer_min <dbl> 10.63, 21.55, 22.80, 12.21, 10.15, 13.31, …
$ EM_nh3_livestock_mm <dbl> 0.2015, 0.2020, 0.2024, 0.2027, 0.2031, 0.…
$ EM_nh3_agr_soils <dbl> 0.1462, 0.1589, 0.1708, 0.1821, 0.1929, 0.…
$ EM_nh3_agr_waste_burn <dbl> 0.0020190, 0.0019560, 0.0018900, 0.0018220…
$ EM_nh3_sum <dbl> 0.6166, 0.6298, 0.6424, 0.6545, 0.6660, 0.…
$ EM_nox_traffic <dbl> 0.8359, 0.8372, 0.8384, 0.8396, 0.8409, 0.…
$ EM_nox_sum <dbl> 1.720, 1.720, 1.720, 1.720, 1.720, 1.721, …
$ EM_so2_sum <dbl> 0.3906, 0.3911, 0.3916, 0.3923, 0.3931, 0.…
$ LI_pigs <dbl> 0.3832, 0.3832, 0.3832, 0.3832, 0.3832, 0.…
$ LI_bovine <dbl> 5.892, 5.892, 5.892, 5.892, 5.892, 5.892, …
$ LI_pigs_v2 <fct> 4.167e-01, 4.167e-01, 4.168e-01, 4.168e-01…
$ LI_bovine_v2 <fct> 6.467e+00, 6.463e+00, 6.458e+00, 6.454e+00…
$ LA_hvi <dbl> 3.998, 3.997, 3.997, 3.997, 3.997, 3.996, …
$ LA_lvi <dbl> 1.234, 1.234, 1.234, 1.234, 1.233, 1.233, …
$ LA_land_use <int> 112, 112, 112, 112, 112, 112, 112, 112, 11…
$ LA_soil_use <int> 17, 17, 17, 17, 17, 17, 17, 17, 17, 17, 17…Ora la variabile Time è di tipo date. Sarà successivamente possibile utilizzare le funzioni year o month per estrarre da ogni data l’anno o il mese.
Il data frame Agrimonia contiene osservazioni giornaliere dal 2016 al 2021:
aria %>%
count(year(Time)) year(Time) n
1 2016 51606
2 2017 51465
3 2018 51465
4 2019 51465
5 2020 51606
6 2021 51465Selezioniamo per comodità le osservazioni riferite al solo anno 2020:
aria2020 = aria %>%
filter(year(Time) == 2020)Il nuovo oggetto aria2020 contiene osservazioni per un numero distinto di giorni dato da
aria2020$Time %>% n_distinct[1] 366e un numero distinto di stazioni di monitoraggio dato da
aria2020$IDStations %>% n_distinct[1] 141Per comodità andremo a rappresentare graficamente solamente la media annuale (2020) delle concentrazioni di PM10 (AQ_pm10) per ogni stazione, la cui posizione è definita dalla coppia Longitudine, Latitudine:
aria2020media = aria2020 %>%
# Seleziono solo le variabili di interesse
select(IDStations, Latitude, Longitude, Time, AQ_pm10) %>%
# Per il calcolo delle medie condizionate ad ogni stazione uso group_by
group_by(IDStations, Latitude, Longitude) %>%
summarise(mediapm = mean(AQ_pm10, na.rm=TRUE))`summarise()` has grouped output by 'IDStations', 'Latitude'. You can override
using the `.groups` argument.glimpse(aria2020media)Rows: 141
Columns: 4
Groups: IDStations, Latitude [141]
$ IDStations <fct> 1264, 1265, 1266, 1269, 1274, 1297, 1374, 1800, 1801, 501, …
$ Latitude <dbl> 46.16785, 45.30278, 45.23349, 45.64970, 46.01583, 45.15047,…
$ Longitude <dbl> 9.879210, 9.495274, 9.666250, 9.601223, 9.286409, 9.930596,…
$ mediapm <dbl> 20.56546, 31.38827, 30.63248, 29.25749, NaN, 37.63559, 29.1…Il nuovo oggetto aria2020media ha 141 righe, come il numero di stazioni. L’opzione na.rm=T permette di calcolare la media anche in presenza di valori mancanti (NA).
E’ già possibile una prima rappresentazione grafica dei dati in aria2020media:
aria2020media %>%
ggplot()+
geom_point(aes(Longitude, Latitude,
col=mediapm)) +
scale_colour_gradient(low="yellow", high="red")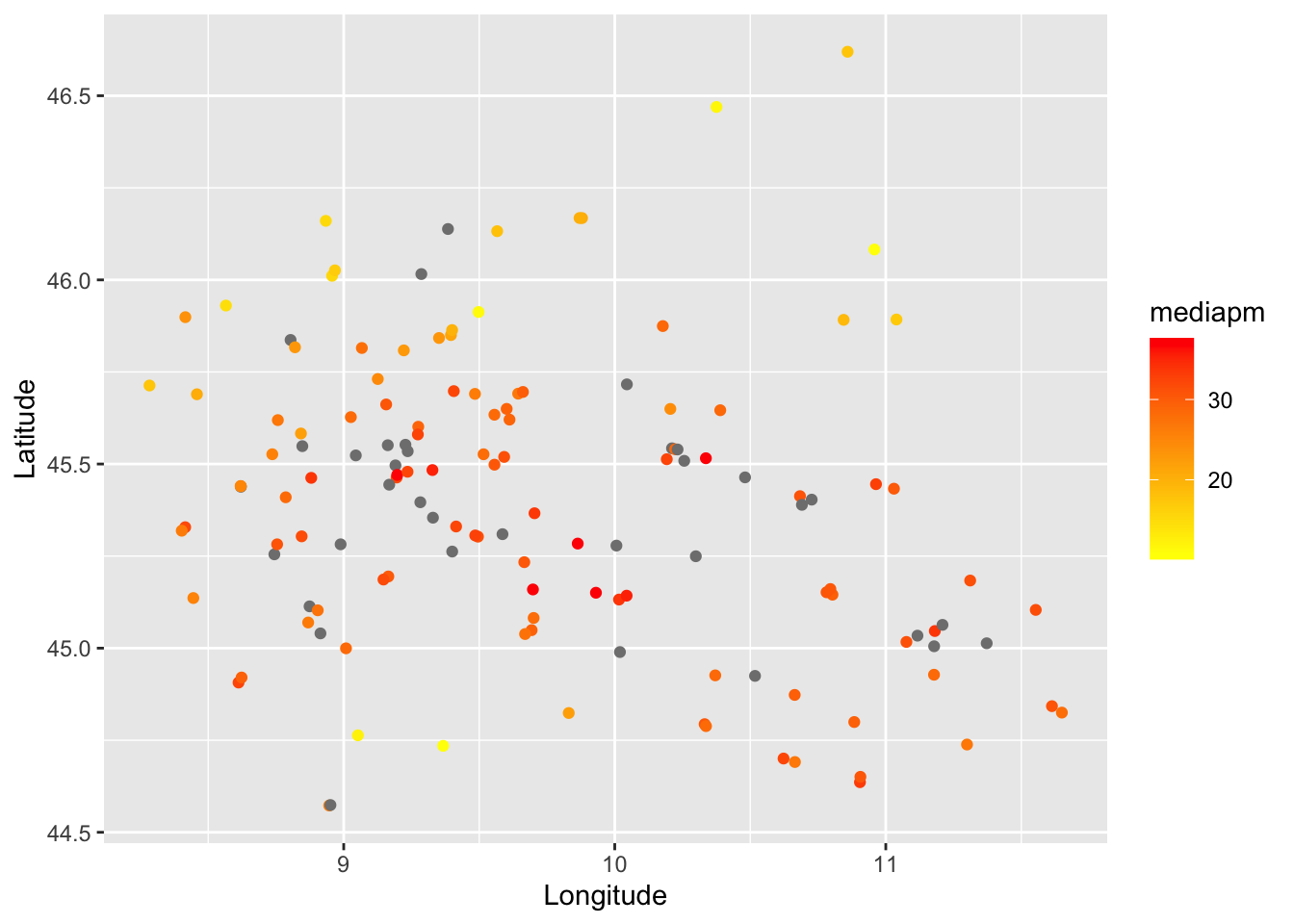
Notare che le coordinate sono di tipo longitudine, latitudine (in gradi).
La scala di colori, con gradiente giallo-rosso, rappresenta la media di PM10 osservata in ogni stazione di monitoraggio. Ogni pallino è una stazione, quelle rappresentate con colore grigio sono quelle che non hanno misurato alcun valore nel 2020 e quindi la media risulta NA. Il problema di questo grafico è che non ha connotazione spaziale, poichè non è stato definito alcun sistema di riferimento delle coordinate.
Procediamo quindi con il trasformare l’oggetto aria2020media in un oggetto di classe sf.
sfSi importi come prima cosa lo shapefile della Lombardia (sistema di coordinate sistema UTMX/Y):
reglomb = st_read("./data/lombardia_com.shp")Reading layer `lombardia_com' from data source
`/Users/michelacameletti/Library/CloudStorage/GoogleDrive-michela.cameletti@unibg.it/My Drive/UniBg/Didattica/Lingue_GeoUrb/2023-2024/MAD2324Rlabs/data/lombardia_com.shp'
using driver `ESRI Shapefile'
Simple feature collection with 1506 features and 12 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 460662.8 ymin: 4947430 xmax: 691489.7 ymax: 5165371
Projected CRS: WGS 84 / UTM zone 32Nclass(reglomb)[1] "sf" "data.frame"La funzione st_as_sf converte un oggetto di tipo data frame in un oggetto spaziale di tipo sf. Questa funzione prende in input l’oggetto da trasformare (aria2020media), i nomi delle variabili che contengono le coordinate (coords=) e il sistema di riferimento (crs) che in questo caso è “WGS84” (da passare tramite la funzione st_crs):
ariasf = st_as_sf(aria2020media,
coords = c("Longitude","Latitude"),
crs = st_crs("WGS84")) %>%
st_transform(st_crs(reglomb))L’ultimo passaggio sopra riportato fa uso della funzione st_transform per fare un cambio di coordinate dal sistema lat/lon del nuovo oggetto spaziale ariasf al sistema UTMX/Y utilizzato dallo shapefile della Lombardia.
Ora che reglomb e ariasf hanno lo stesso sistema di coordinate
st_crs(reglomb) == st_crs(ariasf)[1] TRUEè possibili rappresentarli insieme in uno stesso grafico:
ariasf %>%
ggplot() +
geom_sf(aes(col=mediapm)) +
geom_sf(data=reglomb) +
scale_colour_gradient(low="yellow", high="red")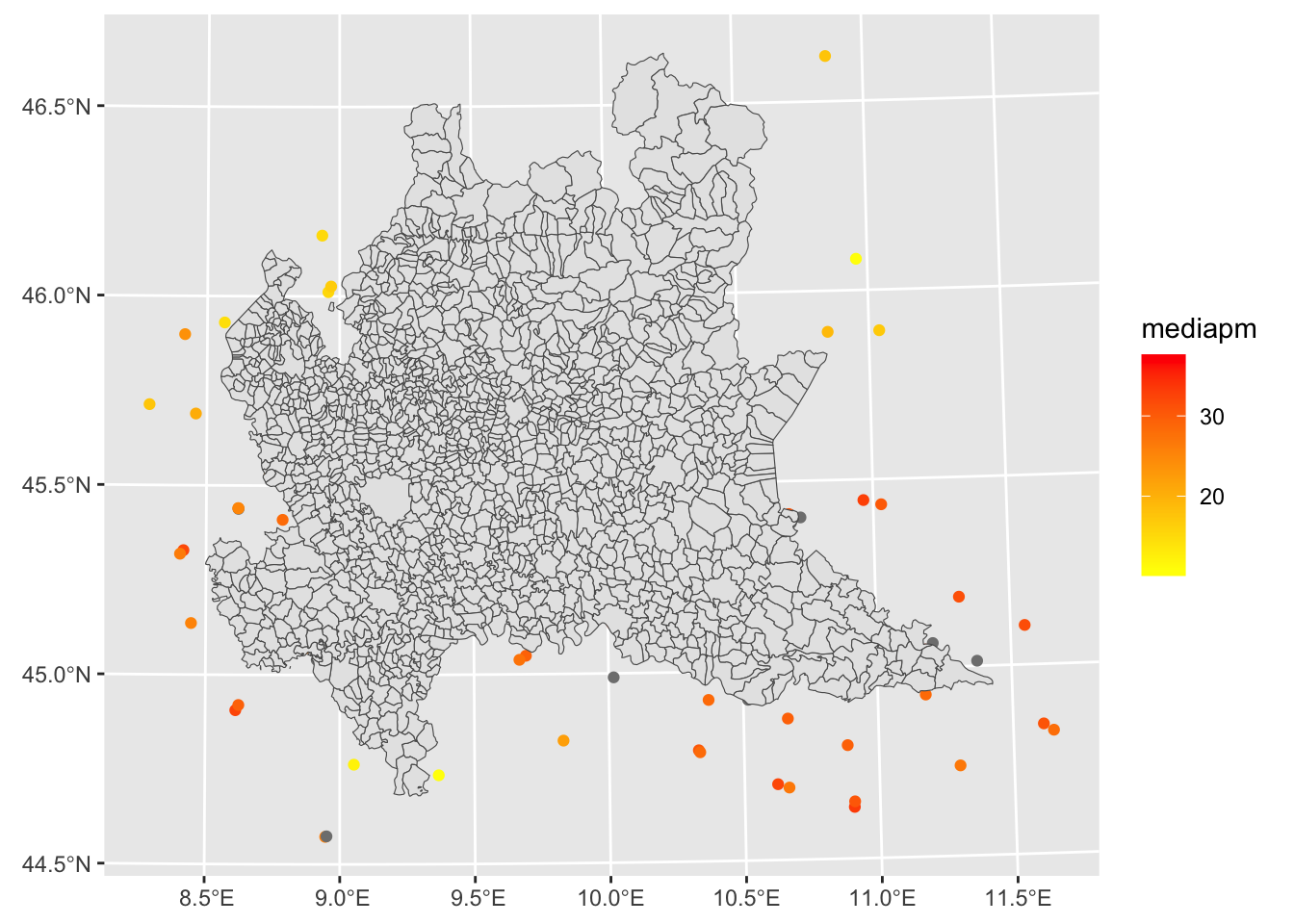
Si notino due cose:
geom_sf (che plotta i punti) prende i dati (mediapm) da ariasf, il secondo geom_sf (che plotta i comuni della Lombardia) prende le informazioni da rappresentare nel grafico da un altro data set (reglomb) che va specificato con data=reglomb.ariasf %>%
ggplot() +
geom_sf(data=reglomb) +
geom_sf(aes(col=mediapm), cex=0.5) +
scale_colour_gradient(low="yellow", high="red")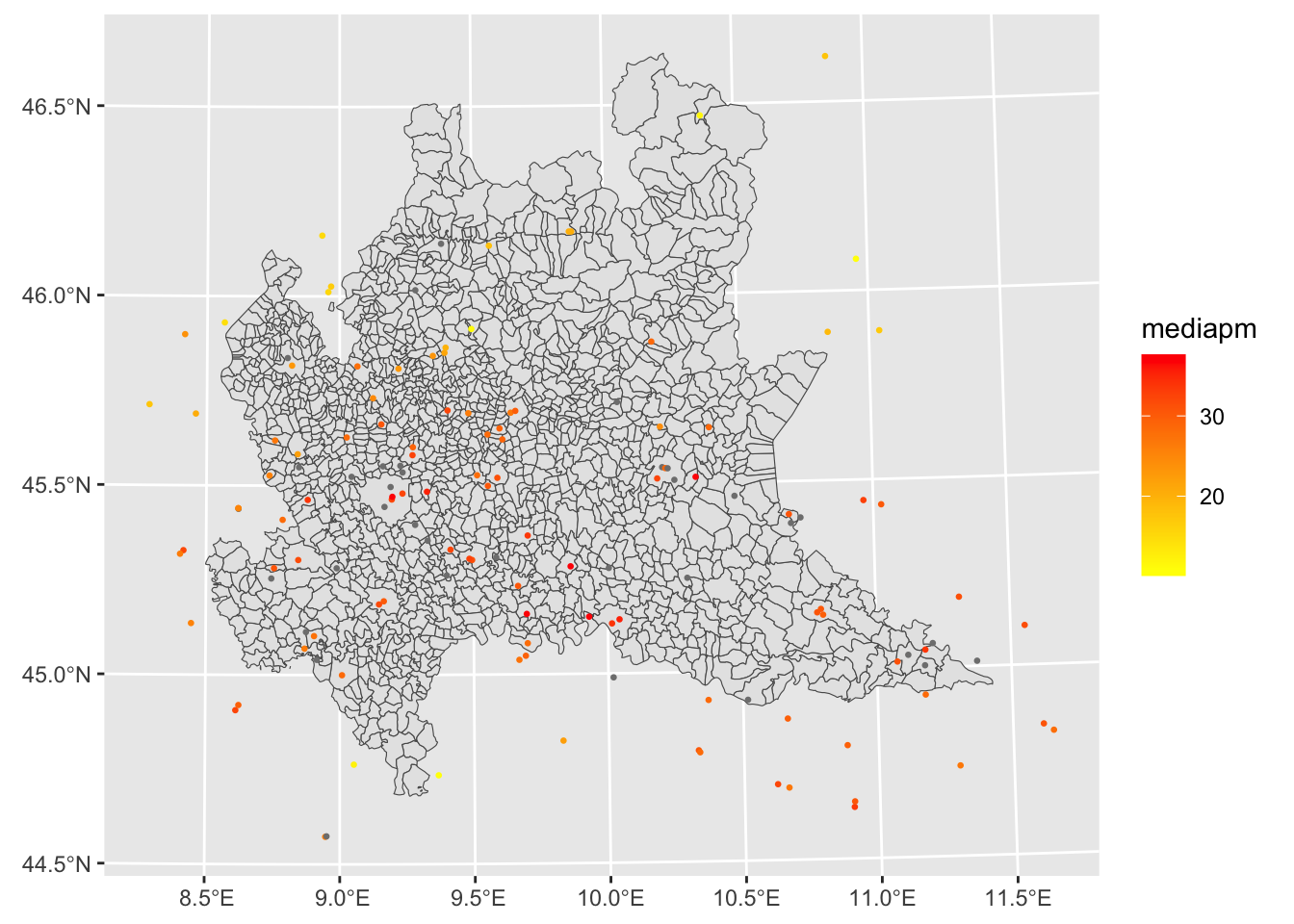
L’opzione cex=0.5 permette di rimpicciolire la dimensione dei punti.
Per una versione interattiva della mappa si può utilizzare la funzione mapview:
ariasf %>%
mapview(zcol="mediapm")Si considerino i dati contenuti nel file pilprocapite.csv relativi al pil procapite (in migliaia di euro) per le regioni italiane per gli anni 2020-2022 (fonte: Istat).
Importare i dati in R creando un oggetto di nome pil. Esplorare il data frame.
Fare il join di pil con lo shapefile (oggestto sf) delle regioni italiane usato nel Laboratorio 7. Creare un nuovo oggetto di tipo sf di nome reg2.
Trasformare reg2 da formato wide a formato long ed eliminare eventuali caratteri non necessari.
Rappresentare graficamente la distribuzione spaziale (mappa) del pil nelle regioni italiane nei 3 anni considerati. Commentare.
Rappresentare graficamente la distribuzione del pil nei 3 anni considerati (senza considerare la dimensione spaziale). Commentare.
Per ogni anno, quale regione ha il pil più elevato? E quale quello minore?
Rappresentare le serie storiche dei valori del pil procapite nel tempo (una linea per ogni regione).
Utilizzare i dati Agrimonia (aria) introdotti nel Laboratorio 7.
Si consideri la variabile WE_temp_2m (temperatura). Rappresentare graficamente la serie storica giornaliera della temperatura (dal 2016 al 2021) per la stazione di monitoraggio 706
Calcolare la temperatura media e mediana separatamente per ogni anno per la stazione 706.
Considerare solo l’ultimo anno disponibile (2021). Calcolare per ogni stazione la temperatura media annua (usare come nome della variabile mediatemp).
Usando case_when (si veda Section 4.4.4) ricodificare la variabile quantitativa continua mediatemp in una variabile categoriale (mediatempcat) con 3 modalità (bassa, media e alta) secondo le seguenti regole:
mediatempcat assume categoria “bassa” se mediatemp è minore di 11 gradi,mediatempcat assume categoria “media” se mediatemp è compresa tra 12 e 14 gradi (si consiglia in questo caso di usare between(mediatemp,12,14)),mediatempcat assume categoria “alta” se mediatemp è maggiore di 14 gradi.La nuova variabile mediatempcat va aggiunta al data frame creato al punto precedente.
mediatempcat) creata al punto precedente.